2020
Motivation
LINCS L1000 데이터는 다양한 변형요인 집합에 의해 유발된 세포발현 데이터를 포함하고 있습니다. 이 데이터는 약물 개발 및 질병의 메커니즘 이해를 위한 귀중한 자원을 제공하지만, 기존의 피크 분해하는 알고리즘은 많은 경우에 유전자의 정확한 발현 수준을 복구할 수 없어 데이터셋에 심각한 노이즈를 유발하며, 생명과학 연구에서의 응용 가능성을 제한합니다.
Result
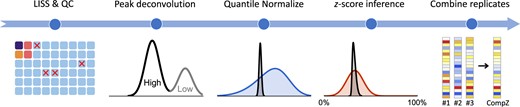
여기서 우리는 peak deconvolution을 위한 베이지안 방법론을 제안하며, 이 방법은 피크의 위치에 대한 unbiased lokelihood estimation을 제공하며, 확률 기반의 z-score를 통해 peak을 활성화 시킨다. 위 알고리즘을 기반으로 우리는 L1000 실험에서의 원시 데이터를 변형 요인의 특징을 나타내는 signature로 처리하기 위한 파이프라인을 구축한다. 제안된 파이프라인의 성능은 biological replicates와 공유된 타겟을 가진 약물 간의 signature 유사성을 기반으로 평가되며, 결과는 우리 파이프라인에서 유래된 signature가 기존의 방법보다 훨씬 신뢰성 있고 유용한 특성을 나타낸다는 것을 보여준다. 따라서 이 새로운 파이프라인은 약물 재활용, 질병 모델링 및 유전자 기능 예측과 같은 응용 분야에서 L1000 자료의 성능을 크게 향상시킬 수 있을 것입니다.
Availability and implementation
LINCS L1000 Phase II (GSE 70138)의 코드와 사전 계산된 데이터는 https://github.com/njpipeorgan/L1000-bayesian 에서 사용 가능하다.