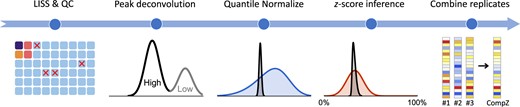
[15] 번째로 읽은 논문의 초록을 다시 보고 정리함. https://jaehong-data.tistory.com/38 [15] L1000CDS2: LINCS L1000 characteristic direction signatures search engine 2016 The library of integrated network-based cellular signatures (LINCS) L1000 데이터 세트는 현재 백만 개 이상의 chemically activated human cell line 유전자 발현 프로필로 구성되어 있다. 고유한 여러 내제 및 외재적인 jaehong-data.tistory.com 초록 LINCS L1000 data set: Chemically perturbated human ..