Tanimoto Coefficient
유사도를 측정하는 metric중 하나이다. Tanimoto coefficient (or similarity)는 화합물 사이의 유사도, 약물 유사도를 측정할때 많이 사용되는 방법이다. 사실 이 유사도는 Jaccard similarity의 continuous version으로, 그냥 Jaccard similarity로 표기하는 사람도 있다. Pearson correlation coefficient 와 point-biserial correlation coefficient 의 관계와 비슷하다.
두 집합의 교집합 크기를, 두 집합의 합집합 크기로 나눈 값으로 정의되며, 식은 아래와 같다.
$$\frac{|A \cap B|}{|A \cup B|}$$
직관적으로 A와 B사이에 얼마나 곂쳤는가가 정량화 된다.
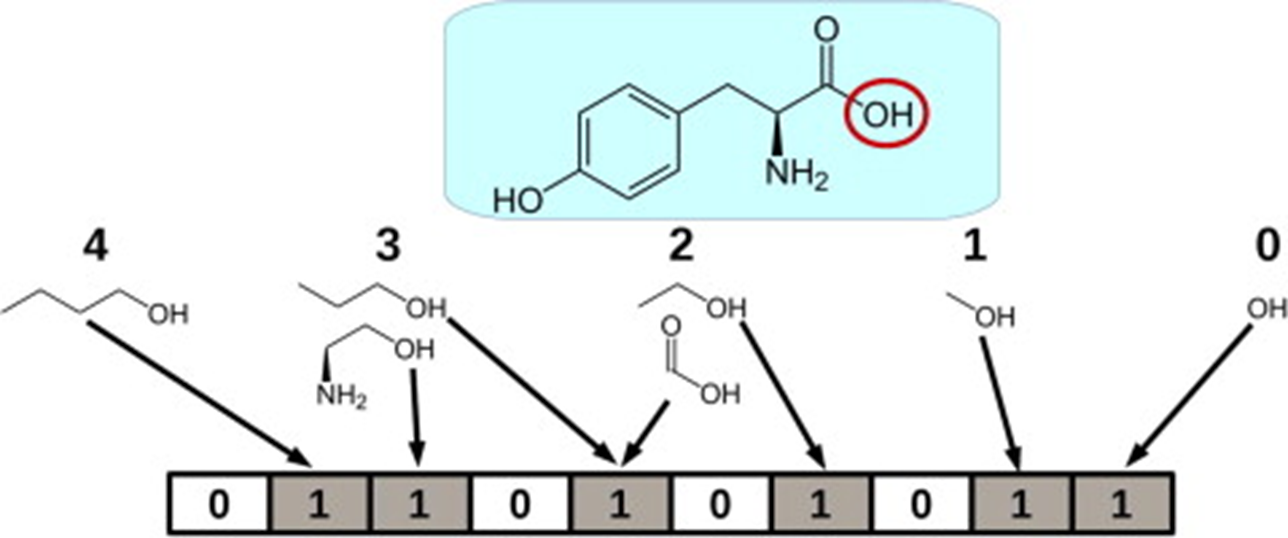
Molecular Fingerprint
화학 구조식간의 유사도를 구하기 위해서는 먼저 molecular fingerprint를 알 필요가 있다. 이는 화학 구조식을 그 고유한 조각들을 0과 1로 인코딩하여 나타내는 벡터이다. 이러한 one-hot 방식은 가장 전통적인 인코딩 방법으로 NLP의 word of bag이 등장하는 count based method와 굉장히 유사하다. 문장이 하나의 molecular 그리고 단어가 하나의 고유한 fragment와 대응된다. 여기서는 더 압축하여 fingerprint를 만드는데, 압축 과정에서 bit-collision이 일어나도 hasing을 잘 하도록 방법론을 제시한다. 그럼에도 NLP에서의 문제점과 마찬가지로 굉장히 sparse한 large matrix가 나오게된다. 이 때문에 distribution based encoding 방법이 나오기도 했다.
Discussion
유사도는 이러한 데이터 구조를 기반으로 Tanimoto index가 간단히 계산된다. 이 외에도 사실 여러 계산 방법이 있지만, reference [1]의 논문에 Tanimoto index가 가장 좋다고 한다.
Outro
Chemical structure embedding은 NLP와 매우 유사한 역사를 가지고 있다. 그렇다면 아직 알아보지는 않았지만, 통계적 방법론도 나오거나, 결국 RNN과 같은 뉴럴 기반의 방법들, 그러다가 어텐션과 트랜스포머까지 가는건 아닌가 싶다.
SSP는 어찌 계산 되는지,
행렬분해 or PCA와는 어떤 연관이 있는지,
component는 몇개를 사용하는게 국룰인지 ... 조사가 필요함
References
[1] https://jcheminf.biomedcentral.com/articles/10.1186/s13321-015-0069-3
Why is Tanimoto index an appropriate choice for fingerprint-based similarity calculations? - Journal of Cheminformatics
Background Cheminformaticians are equipped with a very rich toolbox when carrying out molecular similarity calculations. A large number of molecular representations exist, and there are several methods (similarity and distance metrics) to quantify the simi
jcheminf.biomedcentral.com
[2] https://www.sciencedirect.com/science/article/abs/pii/S1046202314002631
'정리 조금 > Basics' 카테고리의 다른 글
| Molecular Descriptor (0) | 2024.05.02 |
|---|---|
| parquet (0) | 2024.04.22 |
| Deep Copy & Shallow Copy (0) | 2024.03.11 |
| Macro & Micro Averaging (3) | 2024.02.29 |
| Inner & Outer Product (0) | 2024.02.15 |